大岡紘治特任助教と新井宗仁教授(ともに東大総合文化研究科)の研究グループは、タンパク質が特定の構造を作る仕組みを正確に予測する物理学理論を開発した。成果は10月19日付で英科学誌『Nature Communications』に掲載された。
タンパク質は、アミノ酸が平均して数百個つながった高分子。体内ではひも状に伸びた状態で作られるが、アミノ酸配列に応じて折りたたまれて特定の機能を発揮するようになる。タンパク質は生命維持に不可欠であるほか、その特性を生かして医薬品や産業用酵素が開発されているため、タンパク質の立体構造を知ることは重要だ。2020年に開発されたAI「AlphaFold 2」の開発によってタンパク質の構造予測の技術は大きく進歩したが、これはアミノ酸のつながり方をもとに構造を予測するもので、タンパク質が実際に折りたたまれていく仕組みは未解明だった。
一方、1999年には、アミノ酸が100個以下の小型のタンパク質の折りたたみ反応を予測する統計物理学の理論「Wako–Saitô–Muñoz–Eaton(WSME)モデル」が提唱されていた。タンパク質は、分子の持つ「自由エネルギー」という物理量が減少するように折りたたまれる。タンパク質がとる立体構造に対応する自由エネルギーの値を平面に描くことで、自由エネルギーが等しい部分を「等高線」にしてつないだ「自由エネルギー地形」という図を描ける。これによりひも状のタンパク質が自由エネルギーの最も小さい構造をとるまでの経路をたどることができる(図1)。WSMEモデルは、タンパク質が最終的に作る形を指定し、自由エネルギー地形を描けるが、小型のタンパク質の構造しか予測できず、天然に存在するほとんどのタンパク質は大型なので、予測は困難だった。
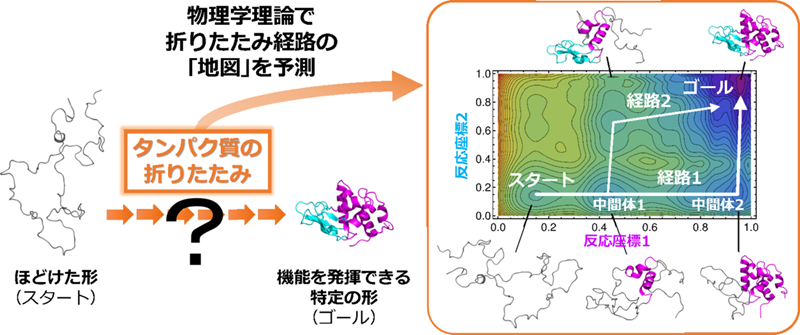
従来のWSMEモデルはタンパク質分子内の隣接するアミノ酸の間に作用する力を重視しており、離れたアミノ酸の間に作用する力は考慮されていなかった。今回の研究では、離れたアミノ酸の間の作用も考慮した新たな理論「WSME-Lモデル」を構築し、厳密解を求めて自由エネルギー地形を効率良く計算できる手法を開発した。検証の結果、従来のWSMEモデルでは正しく予測できなかった大型タンパク質の自由エネルギー地形の予測に成功(図2)。計算時間は小型のタンパク質ではデスクトップPCで約10秒、大型のタンパク質ではスーパーコンピューターで約1時間だった。
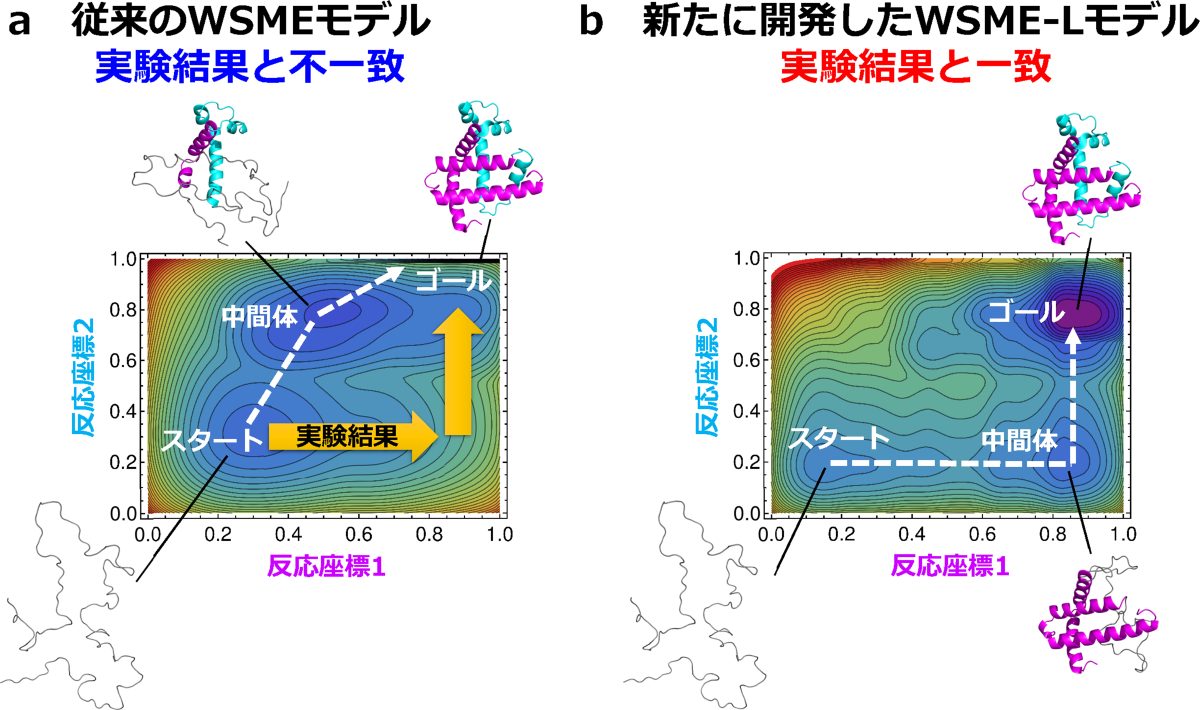
今回開発されたWSME-Lモデルは、多様なタンパク質に対して汎用的に利用可能だ。AlphaFold2の予測などで得られたタンパク質の立体構造にWSME-Lモデルを適用することで、あらゆるタンパク質について折りたたみの仕組みを解明できると期待される。また、得られたデータはタンパク質の動きを予測する新たなAIの開発に活用できる可能性があるほか、医療や工業で用いられる人工タンパク質を大量生産するプロセスの開発や、タンパク質に関係する疾患のメカニズムの解明につながると期待される。
【記事修正】2023年11月6日14時40分 「WSME-Lモデル」の表記が誤っていたため正しく修正しました。











