中川綾哉さん(博士課程・研究当時)、平野央人特任助教、大村紗登士さん(修士課程・研究当時)、濡木理教授(いずれも東大大学院理学系研究科)はCRISPR-Cas酵素の祖先タンパク質TnpBの立体構造を決定した。成果は4月6日付の英国科学雑誌『Nature』に掲載された。
CRISPR(Clustered Regularly Interspaced Short Palindromic Repeats)-Cas酵素は大腸菌など原核生物の獲得免疫に関わる酵素で、特定のDNA配列を切断する活性を持つ。標的となるDNAに相補的な配列を持つRNAをガイドとして利用しており、このRNAをデザインすることで、狙った配列を切断できるゲノム編集ツールとして遺伝子治療などへの応用が進んでいる。Cas酵素には2020年ノーベル化学賞の対象となったCas9をはじめさまざまなタイプが存在し、TnpBは進化してCas12酵素になったと考えられていた。近年の研究によって、TnpBは自身の遺伝子領域内に存在するトランスポゾン(DNA上の位置を転移できる塩基配列)から転写されるノンコーディングRNAと協働し、DNAを切断する活性を持つことが明らかになっていた。ただ、Cas12酵素自体にも種類が複数あり、それぞれどのようにTnpBからCas酵素へ進化したのかなどは分かっていなかった。
中川さんらのグループはクライオ電子顕微鏡を用いて細菌由来のTnpBの構造解析を行った。TnpBのゲノム上のトランスポゾンから転写されたRNAが、その塩基配列からは予測できない複雑な構造をとることでTnpBと複合体を形成していることが明らかになった(図1)。解析した結果、TnpBが12塩基程度の配列を認識し標的のDNAを切断することや、トランスポゾン配列の複製・転移に関与する可能性が分かった。
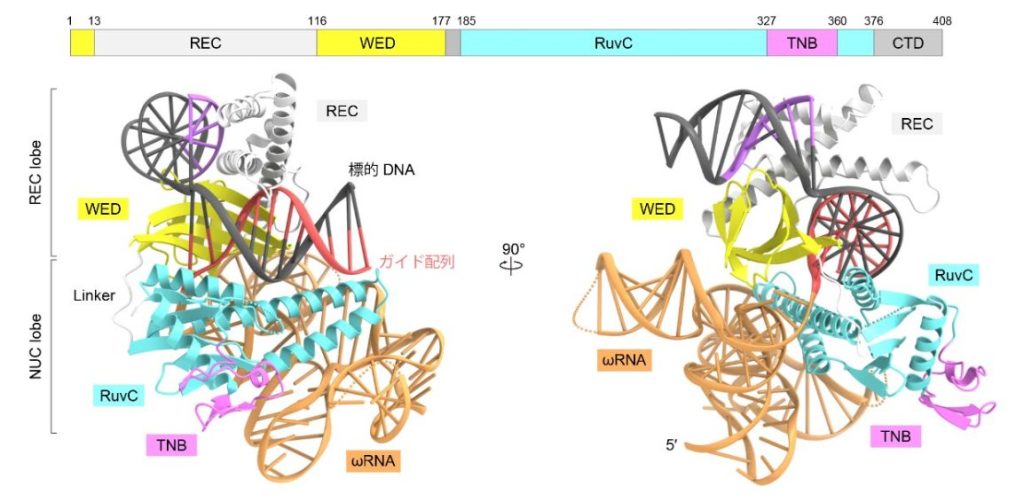
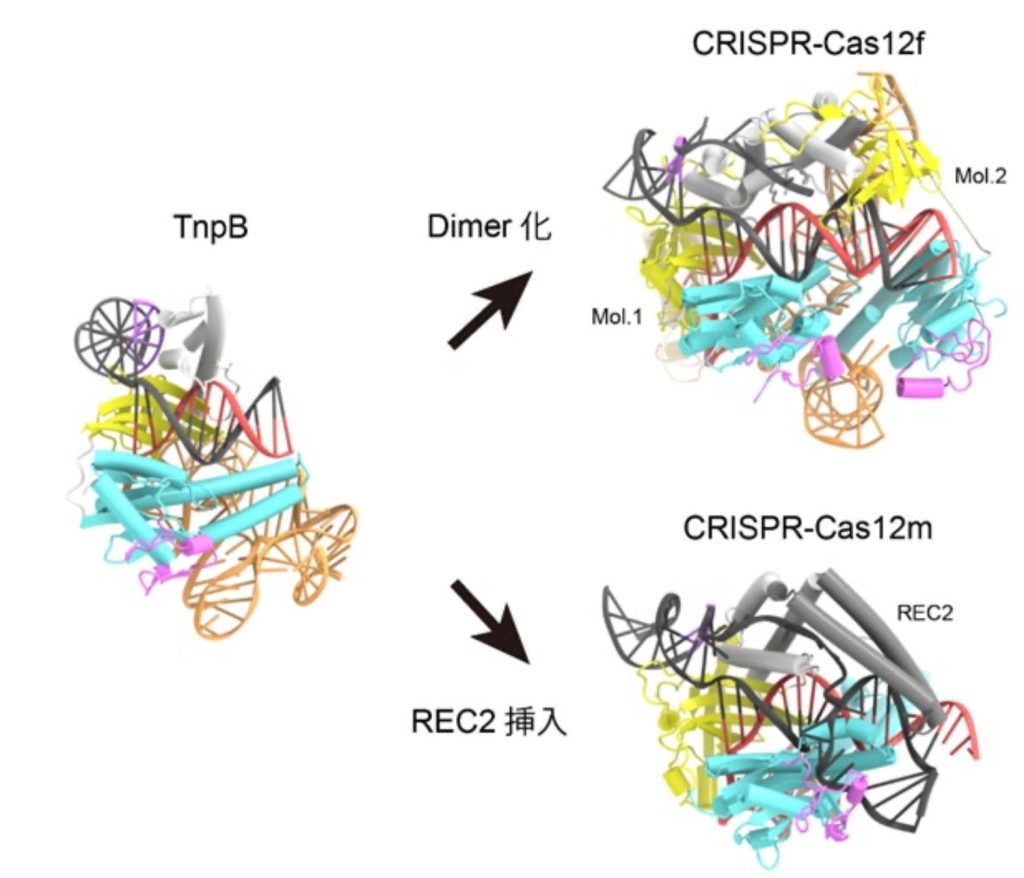
多様なCas12への進化では、TnpBタンパク質から二量体化、新たな機能領域の挿入という異なる過程が関係し、より長い塩基配列を認識する機能を獲得したことも明らかになった(図2)。TnpBの認識できる12塩基配列程度では目的以外の標的DNAを切断する可能性の観点で、免疫機構として不十分。Cas12は長い塩基配列を認識できるようになったことで免疫機構として確立されたと考えられる。成果はCRISPR-Cas獲得免疫機構の理解に貢献し、新規ゲノム編集技術の開発や既存のツールの改良につながると期待される。
【関連記事】










